Una proteina-una struttura-una funzione. È questa la declinazione del paradigma che a partire dal secolo scorso definisce il modo in cui le proteine, le entità operative che presiedono a tutte le reazioni biochimiche cellulari, esplicano la loro attività biologica. Secondo questa teoria, le proteine si ripiegano in una struttura tridimensionale ben definita, dettata dalla loro composizione in amminoacidi, alla quale si associa, in modo univoco, una funzione. Questa forma è responsabile ad esempio del corretto funzionamento degli enzimi, i catalizzatori delle reazioni biochimiche, perché permette loro di riconoscere selettivamente i rispettivi substrati. Questi interagiscono in modo complementare con la proteina così come una chiave entra nella propria serratura.
Eppure, nella mia attività di ricerca, ho incontrato un enzima che non obbedisce al paradigma struttura-funzione. Questa proteina si chiama UreG ed è coinvolta nell’attivazione dell’ureasi, un fattore di patogenicità di alcuni agenti infettivi, quali, ad esempio, il batterio dell’ulcera Helicobacter pylori. Studiando questo enzima, nel nostro gruppo di ricerca del Dipartimento di Farmacia e Biotecnologie all’Università di Bologna, avevamo scoperto che UreG in soluzione non assume una conformazione ben definita, ma esiste invece in un insieme conformazionale costituito da tante strutture diverse in equilibrio tra loro. Queste osservazioni ci avevano permesso di attribuire UreG alla famiglia delle proteine intrinsecamente disordinate (intrinsically disordered proteins, IDP). Queste proteine funzionano correttamente senza assumere una rigida struttura tridimensionale, e la loro scoperta ha portato, negli ultimi quindici anni, alla ridefinizione della validità generale del paradigma struttura-funzione. In questo stato parzialmente disordinato, UreG mostra una certa funzionalità, essendo cataliticamente attivo.
Qual è la relazione tra la flessibilità strutturale di UreG e la sua attività catalitica? Per rispondere a questa domanda, abbiamo condotto uno studio sperimentale, i cui risultati sono stati pubblicati su Scientific Reports. In base alla relazione struttura-funzione ci saremmo aspettati che, spostando l’equilibrio conformazionale della proteina verso uno stato più rigido e ordinato, la sua attività enzimatica aumentasse di pari passo.
Per verificarlo, abbiamo perturbato la conformazione della proteina aggiungendo in soluzione molecole capaci di aumentare o di diminuire il disordine strutturale, e abbiamo osservato il loro effetto sia sulla struttura della proteina sia sulla sua funzionalità. Poiché le tecniche convenzionali ad alta risoluzione, come la cristallografia di raggi X, nate per studiare proteine con una struttura rigida, spesso sono inadeguate per caratterizzare le IDP, per studiare la conformazione di questo enzima abbiamo usato un diverso approccio sperimentale. Attraverso la collaborazione con Elisabetta Mileo del CNRS di Marsiglia, abbiamo utilizzato una tecnica chiamata risonanza paramagnetica elettronica, che definisce la mobilità della proteina in soluzione, e la correla con il suo livello di disordine strutturale. Parallelamente, a Bologna, abbiamo completato l’analisi strutturale della proteina e abbiamo studiato la sua funzionalità nelle stesse condizioni sperimentali attraverso saggi enzimatici.
I dati ottenuti ci hanno dimostrato una situazione ben diversa rispetto a quanto atteso: lo spostamento conformazionale della proteina verso una maggiore rigidità, infatti, causa la totale perdita della sua attività enzimatica. In altre parole, questo enzima ha bisogno di esistere in una finestra di flessibilità strutturale per funzionare correttamente, e, di conseguenza, la sua rigidità strutturale non è direttamente correlata alla sua funzione (Figura 1).
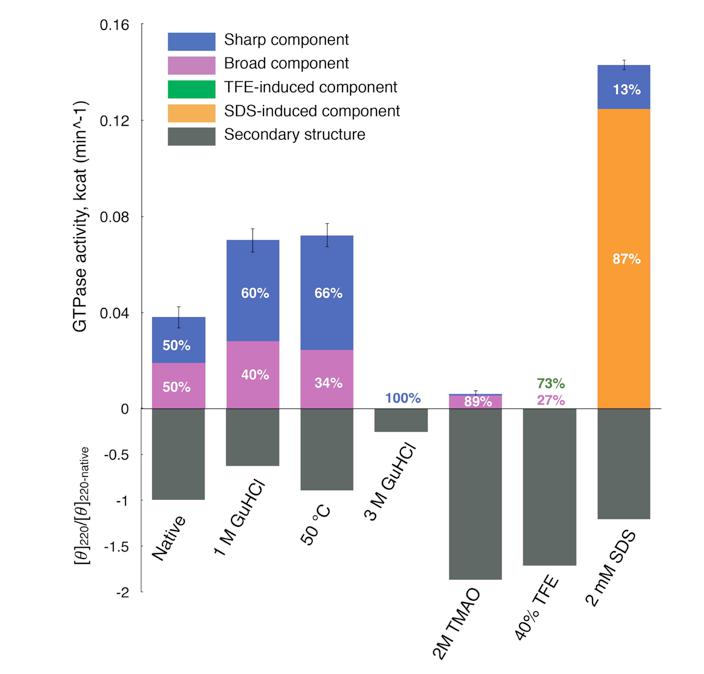
Figura 1. L’istogramma indica la mancata correlazione tra struttura e attività enzimatica per UreG (da Palombo et al. Scientific Reports, 2017)
Il fatto che alcuni enzimi non obbediscano al paradigma struttura-funzione ci porta a fare alcune considerazioni. La prima, di carattere generale, riguarda il possibile il ruolo del disordine strutturale per l’attività enzimatica. Per gli enzimi intrinsecamente disordinati come UreG, la flessibilità della forma potrebbe costituire un meccanismo di regolazione per consentire alla proteina di essere cataliticamente attiva solo nell’ambiente cellulare giusto, o nell’interazione con i partner molecolari corretti. Il disordine quindi non sarebbe casuale e incontrollato, ma sarebbe invece una precisa strategia di regolazione di alcune reazioni biochimiche.
La seconda considerazione, di carattere applicativo, riguarda invece possibili nuove strategie sperimentali nella ricerca farmaceutica, che oggi basa il suo razionale sullo studio di inibitori enzimatici visti secondo il modello della chiave-serratura. Questo studio suggerisce che, in alcuni casi, invece di ricercare molecole che mimino la chiave specifica che interagisce con l’enzima, l’inibizione funzionale possa passare attraverso il blocco della mobilità conformazionale della proteina target.
Riferimenti: Scientific Reports